UTILIZA SARS-COV-2 ESTRATEGIA MOLECULAR INUSUAL, PERO EFICAZ PARA ENTRAR A CÉLULAS
12 julio, 2020El nuevo coronavirus evolucionó en la naturaleza y se adaptó para infectar.
Fuente: Cinvestav
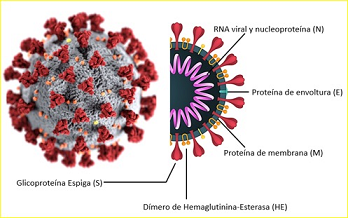
Luis Delaye Arredondo, investigador del Departamento de Ingeniería Genética del Centro de Investigación y Estudios Avanzados (Cinvestav) Irapuato, señaló que si bien, en redes sociales y sitios de internet circula la teoría de que el SARS-CoV-2 fue creado con el propósito de usarlo contra la población, uno de los datos que respaldan su origen natural está relacionado con la proteína espiga o S, que el virus utiliza para entrar a las células humanas.
En caso de que el SARS-CoV-2 hubiera sido diseñado en un laboratorio, su proteína S utilizaría una estrategia molecular óptima ya identificada por los científicos en el 2012. En cambio, la que este virus emplea es nueva; lo cual sugiere que evolucionó en la naturaleza. Tras entrar al organismo, el nuevo coronavirus se une a células específicas del epitelio respiratorio a través de la proteína S. Ésta se encuentra en la superficie del virus y es capaz de reconocer a la proteína llamada enzima convertidora de angiotensina (ACE2 por sus siglas en inglés), que se encuentra en la parte externa de las células humanas, y es necesaria en el proceso de regulación de la presión sanguínea.
La interacción de la proteína S con las células humanas es similar al de una llave y una cerradura, por lo cual debe existir similitud entre la forma de ambas para que el mecanismo funcione: la puerta se abra y el virus pueda entrar. Para estudiar la interacción entre la proteína S y la ACE2, diversos grupos de investigación se han enfocado en algunos coronavirus que infectan a los humanos y a otros mamíferos, entre ellos el murciélago, el pangolín y la civeta.
Lo anterior con el objetivo de identificar la estructura que debería tener la proteína S para reconocer a la proteína humana ACE2 de manera eficiente. En específico han estudiado el segmento conocido como RBD (las siglas en inglés de Receptor Binding Domain), que los coronavirus usan para interactuar con una célula y el cual equivale a los dientes de la llave. Este segmento de la proteína S contiene cinco aminoácidos, que son cruciales pare el reconocimiento de la ACE2, y pueden ser distintos entre los coronavirus que infectan a diversas especies.
Al comparar los distintos aminoácidos de estos coronavirus, mediante programas bioinformáticos y pruebas experimentales, en el 2012 se identificó la combinación que resultaría más eficiente para interactuar con la proteína ACE2 humana. Recientemente, el análisis del SARS-CoV-2 arrojó que su secuencia coincide con la descrita como óptima en solo un aminoácido, lo anterior llevó a pensar que la afinidad de su proteína S con la ACE2 humana sería baja, pero los estudios experimentales han demostrado que logra reconocerla eficientemente.
“Diseñar esta nueva combinación de aminoácidos, hubiera requerido un trabajo experimental enorme para los científicos, y esto hace más probable que el SARS-CoV-2 sea resultado de un proceso evolutivo”.
Una explicación de lo anterior, es que el SARS-CoV-2 evolucionó a través de la recombinación de coronavirus que infectan tanto a murciélagos como a pangolines. Y aunque no se sabe con certeza qué especie animal le transfirió el nuevo coronavirus al humano, es posible que el hospedero intermediario tuviera una proteína similar a la ACE2 humana.
Entonces, para que un virus de origen animal logre infectar al humano, no siempre requiere de un gran número de mutaciones, de ahí la necesidad de monitorear las variantes genéticas que aparezcan en la población conforme la pandemia avanza y el SARS-CoV-2 evoluciona, en especial porque algunos cambios en su material genético impactan en su funcionamiento, por ejemplo en su mayor o menor agresividad.

